| 75 > Pert% ≤ 100 | |
| 50 > Pert% ≤ 75 | |
| 25 > Pert% ≤ 50 | |
| 0 > Pert% ≤ 25 | |
| Pert% = 0 | |
| -25 ≥ Pert% < 0 | |
| -50 ≥ Pert% < -25 | |
| -75 ≥ Pert% < -50 | |
| -100 ≥ Pert% < -75 |
| Index | Node1 | Node2 | Owner | 3lnx Freq | 3lny Freq | 3lnx Hub1? | 3lny Hub1? | 3lnx Hub2? | 3lny Hub2? | Value1 | Value2 |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | A:A:3 | A:A:91 | 3lnx | 75.56 | 0 | No | No | Yes | Yes | 0 | 0 |
| 2 | A:A:13 | A:A:14 | 3lnx | 55.92 | 48.97 | Yes | Yes | No | No | 0 | 0 |
| 3 | A:A:22 | A:A:71 | 3lnx | 56.64 | 0 | No | No | No | Yes | 0 | 0 |
| 4 | A:A:52 | A:A:53 | 3lnx | 67.55 | 0 | No | No | Yes | No | 0 | 0 |
| 5 | A:A:57 | A:A:59 | 3lnx | 76.83 | 0 | Yes | Yes | No | No | 0 | 0 |
| 6 | A:A:57 | A:A:90 | 3lnx | 59.56 | 33.11 | Yes | Yes | No | No | 0 | 0 |
| 7 | A:A:76 | A:A:79 | 3lnx | 55.58 | 0 | Yes | No | No | No | 0 | 0 |
| 8 | A:A:10 | A:A:9 | 3lnx | 0 | 0 | No | No | No | No | 0 | 0 |
| 9 | A:A:13 | A:A:83 | 3lnx | 40.01 | 0 | Yes | Yes | No | No | 0 | 0 |
| 10 | A:A:60 | A:A:61 | 3lnx | 0.09 | 0 | No | No | Yes | No | 0 | 0 |
| 11 | A:A:34 | A:A:36 | 3lnx | 17.83 | 0 | No | No | Yes | Yes | 0 | 0 |
| 12 | A:A:11 | A:A:18 | 3lnx | 0 | 0 | No | No | No | No | 0 | 0 |
| 13 | A:A:25 | A:A:35 | 3lnx | 0 | 0 | No | No | No | No | 0 | 0 |
| 14 | A:A:45 | A:A:48 | 3lnx | 0 | 0 | No | No | No | No | 0 | 0 |
| 15 | A:A:53 | A:A:94 | 3lnx | 16.4 | 0 | Yes | No | No | No | 0 | 0 |
| 16 | A:A:1 | A:A:2 | Shared | 100 | 99.99 | No | No | No | No | 0 | 0 |
| 17 | A:A:2 | A:A:3 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 18 | A:A:2 | A:A:5 | Shared | 90.28 | 26.27 | No | No | No | No | 0 | 0 |
| 19 | A:A:3 | A:A:4 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 20 | A:A:5 | A:A:6 | Shared | 99.89 | 100 | No | No | No | No | 0 | 0 |
| 21 | A:A:6 | A:A:7 | Shared | 97.25 | 98.6 | No | No | Yes | Yes | 0 | 0 |
| 22 | A:A:7 | A:A:8 | Shared | 67.53 | 72.56 | Yes | Yes | No | No | 0 | 0 |
| 23 | A:A:7 | A:A:89 | Shared | 99.98 | 100 | Yes | Yes | No | No | 0 | 0 |
| 24 | A:A:7 | A:A:91 | Shared | 78.42 | 64.62 | Yes | Yes | Yes | Yes | 0 | 0 |
| 25 | A:A:8 | A:A:9 | Shared | 99.86 | 99.93 | No | No | No | No | 0 | 0 |
| 26 | A:A:8 | A:A:86 | Shared | 87.6 | 91.18 | No | No | Yes | Yes | 0 | 0 |
| 27 | A:A:86 | A:A:9 | Shared | 58.55 | 51.17 | Yes | Yes | No | No | 0 | 0 |
| 28 | A:A:87 | A:A:9 | Shared | 83.67 | 68.05 | No | No | No | No | 0 | 0 |
| 29 | A:A:10 | A:A:11 | Shared | 96.75 | 71.38 | No | No | No | No | 0 | 0 |
| 30 | A:A:13 | A:A:81 | Shared | 98.37 | 99.32 | Yes | Yes | No | No | 0 | 0 |
| 31 | A:A:14 | A:A:15 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 32 | A:A:15 | A:A:16 | Shared | 99.37 | 99.75 | No | No | No | No | 0 | 0 |
| 33 | A:A:17 | A:A:18 | Shared | 93.61 | 95.31 | No | No | No | No | 0 | 0 |
| 34 | A:A:20 | A:A:21 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 35 | A:A:21 | A:A:22 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 36 | A:A:22 | A:A:23 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 37 | A:A:23 | A:A:24 | Shared | 98.03 | 92.89 | No | No | No | No | 0 | 0 |
| 38 | A:A:26 | A:A:27 | Shared | 83.22 | 69.48 | No | No | No | No | 0 | 0 |
| 39 | A:A:27 | A:A:28 | Shared | 100 | 99.93 | No | No | No | No | 0 | 0 |
| 40 | A:A:28 | A:A:29 | Shared | 81.15 | 88.1 | No | No | No | No | 0 | 0 |
| 41 | A:A:29 | A:A:30 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 42 | A:A:30 | A:A:31 | Shared | 99.97 | 100 | No | No | No | No | 0 | 0 |
| 43 | A:A:31 | A:A:32 | Shared | 78.65 | 89.18 | No | No | No | No | 0 | 0 |
| 44 | A:A:31 | A:A:36 | Shared | 53.53 | 75.75 | No | No | Yes | Yes | 0 | 0 |
| 45 | A:A:35 | A:A:36 | Shared | 100 | 100 | No | No | Yes | Yes | 0 | 0 |
| 46 | A:A:35 | A:A:58 | Shared | 89.13 | 52.03 | No | No | No | No | 0 | 0 |
| 47 | A:A:36 | A:A:37 | Shared | 100 | 100 | Yes | Yes | No | No | 0 | 0 |
| 48 | A:A:37 | A:A:38 | Shared | 98.26 | 97.49 | No | No | No | No | 0 | 0 |
| 49 | A:A:40 | A:A:41 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 50 | A:A:41 | A:A:42 | Shared | 100 | 100 | No | No | Yes | Yes | 0 | 0 |
| 51 | A:A:42 | A:A:43 | Shared | 99.47 | 99.61 | Yes | Yes | No | No | 0 | 0 |
| 52 | A:A:42 | A:A:47 | Shared | 89.55 | 71.76 | Yes | Yes | No | No | 0 | 0 |
| 53 | A:A:47 | A:A:48 | Shared | 97.18 | 97.58 | No | No | No | No | 0 | 0 |
| 54 | A:A:48 | A:A:49 | Shared | 86.98 | 90.94 | No | No | No | No | 0 | 0 |
| 55 | A:A:49 | A:A:51 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 56 | A:A:51 | A:A:52 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 57 | A:A:52 | A:A:89 | Shared | 57.65 | 58.72 | No | No | No | No | 0 | 0 |
| 58 | A:A:53 | A:A:54 | Shared | 100 | 100 | Yes | No | No | No | 0 | 0 |
| 59 | A:A:53 | A:A:56 | Shared | 74.84 | 62.86 | Yes | No | No | No | 0 | 0 |
| 60 | A:A:56 | A:A:91 | Shared | 94.54 | 100 | No | No | Yes | Yes | 0 | 0 |
| 61 | A:A:57 | A:A:58 | Shared | 100 | 100 | Yes | Yes | No | No | 0 | 0 |
| 62 | A:A:58 | A:A:59 | Shared | 99.84 | 99.86 | No | No | No | No | 0 | 0 |
| 63 | A:A:61 | A:A:62 | Shared | 99.99 | 100 | Yes | No | No | No | 0 | 0 |
| 64 | A:A:61 | A:A:64 | Shared | 99.71 | 99.82 | Yes | No | No | No | 0 | 0 |
| 65 | A:A:64 | A:A:65 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 66 | A:A:65 | A:A:66 | Shared | 91.58 | 85.93 | No | No | No | No | 0 | 0 |
| 67 | A:A:66 | A:A:67 | Shared | 91.3 | 95.58 | No | No | No | No | 0 | 0 |
| 68 | A:A:70 | A:A:71 | Shared | 99.99 | 100 | No | No | No | Yes | 0 | 0 |
| 69 | A:A:71 | A:A:72 | Shared | 99.74 | 100 | No | Yes | No | No | 0 | 0 |
| 70 | A:A:72 | A:A:73 | Shared | 99.72 | 100 | No | No | No | No | 0 | 0 |
| 71 | A:A:75 | A:A:76 | Shared | 100 | 99.99 | No | No | Yes | No | 0 | 0 |
| 72 | A:A:76 | A:A:77 | Shared | 100 | 100 | Yes | No | No | No | 0 | 0 |
| 73 | A:A:76 | A:A:80 | Shared | 54.99 | 63.78 | Yes | No | No | No | 0 | 0 |
| 74 | A:A:77 | A:A:78 | Shared | 99.41 | 99.12 | No | No | No | No | 0 | 0 |
| 75 | A:A:78 | A:A:79 | Shared | 98.4 | 95.65 | No | No | No | No | 0 | 0 |
| 76 | A:A:79 | A:A:80 | Shared | 90.1 | 97.12 | No | No | No | No | 0 | 0 |
| 77 | A:A:80 | A:A:81 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 78 | A:A:83 | A:A:84 | Shared | 92.1 | 99.49 | No | No | No | No | 0 | 0 |
| 79 | A:A:84 | A:A:85 | Shared | 100 | 100 | No | No | No | No | 0 | 0 |
| 80 | A:A:85 | A:A:86 | Shared | 77.98 | 59.62 | No | No | Yes | Yes | 0 | 0 |
| 81 | A:A:86 | A:A:87 | Shared | 99.93 | 99.93 | Yes | Yes | No | No | 0 | 0 |
| 82 | A:A:88 | A:A:89 | Shared | 99.96 | 99.96 | No | No | No | No | 0 | 0 |
| 83 | A:A:90 | A:A:91 | Shared | 99.99 | 99.99 | No | No | Yes | Yes | 0 | 0 |
| 84 | A:A:93 | A:A:94 | Shared | 83.91 | 64.71 | No | No | No | No | 0 | 0 |
| 85 | A:A:23 | A:A:36 | Shared | 27.05 | 0 | No | No | Yes | Yes | 0 | 0 |
| 86 | A:A:30 | A:A:33 | Shared | 30.16 | 10.48 | No | No | No | No | 0 | 0 |
| 87 | A:A:91 | A:A:92 | Shared | 20.33 | 0.08 | Yes | Yes | No | No | 0 | 0 |
| 88 | A:A:49 | A:A:50 | Shared | 24.27 | 24.14 | No | No | No | No | 0 | 0 |
| 89 | A:A:81 | A:A:82 | Shared | 0.77 | 0.6 | No | No | No | No | 0 | 0 |
| 90 | A:A:61 | A:A:63 | Shared | 0 | 0 | Yes | No | No | No | 0 | 0 |
| 91 | A:A:42 | A:A:44 | Shared | 0 | 0 | Yes | Yes | No | No | 0 | 0 |
| 92 | A:A:54 | A:A:55 | Shared | 0.09 | 0.14 | No | No | No | No | 0 | 0 |
| 93 | A:A:18 | A:A:19 | Shared | 0 | 0 | No | No | No | No | 0 | 0 |
| 94 | A:A:74 | A:A:75 | Shared | 1.51 | 1.65 | No | No | No | No | 0 | 0 |
| 95 | A:A:69 | A:A:70 | Shared | 0.03 | 0 | No | No | No | No | 0 | 0 |
| 96 | A:A:67 | A:A:68 | Shared | 0.22 | 3.24 | No | No | No | No | 0 | 0 |
| 97 | A:A:21 | A:A:39 | Shared | 0 | 0 | No | No | No | No | 0 | 0 |
| 98 | A:A:12 | A:A:13 | Shared | 0 | 0 | No | No | Yes | Yes | 0 | 0 |
| 99 | A:A:60 | A:A:88 | Shared | 0.04 | 0.56 | No | No | No | No | 0 | 0 |
| 100 | A:A:46 | A:A:47 | Shared | 0 | 0 | No | No | No | No | 0 | 0 |
| 101 | A:A:11 | A:A:85 | 3lny | 45.26 | 64.18 | No | No | No | No | 0 | 0 |
| 102 | A:A:13 | A:A:16 | 3lny | 43.78 | 56.17 | Yes | Yes | No | No | 0 | 0 |
| 103 | A:A:20 | B:B:8 | 3lny | 0 | 88.22 | No | No | No | No | 0 | 0 |
| 104 | A:A:22 | B:B:6 | 3lny | 0 | 96.5 | No | No | No | No | 0 | 0 |
| 105 | A:A:36 | A:A:57 | 3lny | 0 | 60.54 | Yes | Yes | Yes | Yes | 0 | 0 |
| 106 | A:A:51 | A:A:91 | 3lny | 0 | 85.04 | No | No | Yes | Yes | 0 | 0 |
| 107 | A:A:71 | A:A:75 | 3lny | 0 | 74.5 | No | Yes | No | No | 0 | 0 |
| 108 | A:A:71 | B:B:4 | 3lny | 0 | 96.81 | No | Yes | No | No | 0 | 0 |
| 109 | A:A:71 | B:B:5 | 3lny | 0 | 52.14 | No | Yes | No | No | 0 | 0 |
| 110 | B:B:3 | B:B:4 | 3lny | 0 | 55.13 | No | No | No | No | 0 | 0 |
| 111 | B:B:4 | B:B:5 | 3lny | 0 | 91.48 | No | No | No | No | 0 | 0 |
| 112 | B:B:5 | B:B:6 | 3lny | 0 | 100 | No | No | No | No | 0 | 0 |
| 113 | A:A:17 | B:B:8 | 3lny | 0 | 0 | No | No | No | No | 0 | 0 |
| 114 | A:A:56 | A:A:57 | 3lny | 0 | 0 | No | No | Yes | Yes | 0 | 0 |
| 115 | A:A:57 | A:A:67 | 3lny | 0 | 38.02 | Yes | Yes | No | No | 0 | 0 |
| 116 | A:A:34 | A:A:57 | 3lny | 0 | 22.43 | No | No | Yes | Yes | 0 | 0 |
| 117 | B:B:6 | B:B:7 | 3lny | 0 | 0 | No | No | No | No | 0 | 0 |
| 118 | A:A:24 | A:A:25 | 3lny | 0 | 0 | No | No | No | No | 0 | 0 |
| 119 | A:A:45 | A:A:46 | 3lny | 0 | 0 | No | No | No | No | 0 | 0 |
| 120 | A:A:4 | A:A:93 | 3lny | 0 | 11.01 | No | No | No | No | 0 | 0 |
Index: link id, click on each number to highlight the corresponding link in the 3D visualization.
Node1 Node2: the two nodes of the corresponding link.
Owner: the name of the network the link belong to or "Shared" if the corresponding link is present in both networks.
Freq: the trajectory frequency of this link or 0 if the link is not present.
Hub1?, Hub2?: "Yes" if the corresponding node has more than 3 links, otherwise "No".
Value1, Value2: these columns report the values present in the user submitted external values file (if any).
| Index | Node1 | Node2 | 3lnx Freq | 3lny Freq | Freq Pert | Value1 | Value2 |
|---|---|---|---|---|---|---|---|
| 1 | A:A:1 | A:A:2 | 100 | 99.99 | -0.0100021 | 0 | 0 |
| 2 | A:A:2 | A:A:3 | 100 | 100 | -0 | 0 | 0 |
| 3 | A:A:2 | A:A:5 | 90.28 | 26.27 | -70.9016 | 0 | 0 |
| 4 | A:A:3 | A:A:4 | 100 | 100 | -0 | 0 | 0 |
| 5 | A:A:3 | A:A:91 | 75.56 | 12.73 | -83.1525 | 0 | 0 |
| 6 | A:A:5 | A:A:6 | 99.89 | 100 | 0.110001 | 0 | 0 |
| 7 | A:A:6 | A:A:7 | 97.25 | 98.6 | 1.36917 | 0 | 0 |
| 8 | A:A:7 | A:A:8 | 67.53 | 72.56 | 6.93219 | 0 | 0 |
| 9 | A:A:7 | A:A:89 | 99.98 | 100 | 0.0199966 | 0 | 0 |
| 10 | A:A:7 | A:A:91 | 78.42 | 64.62 | -17.5975 | 0 | 0 |
| 11 | A:A:8 | A:A:9 | 99.86 | 99.93 | 0.0700487 | 0 | 0 |
| 12 | A:A:8 | A:A:86 | 87.6 | 91.18 | 3.9263 | 0 | 0 |
| 13 | A:A:86 | A:A:9 | 58.55 | 51.17 | -12.6046 | 0 | 0 |
| 14 | A:A:87 | A:A:9 | 83.67 | 68.05 | -18.6686 | 0 | 0 |
| 15 | A:A:10 | A:A:11 | 96.75 | 71.38 | -26.2222 | 0 | 0 |
| 16 | A:A:13 | A:A:14 | 55.92 | 48.97 | -12.4285 | 0 | 0 |
| 17 | A:A:13 | A:A:81 | 98.37 | 99.32 | 0.956501 | 0 | 0 |
| 18 | A:A:14 | A:A:15 | 100 | 100 | -0 | 0 | 0 |
| 19 | A:A:15 | A:A:16 | 99.37 | 99.75 | 0.38095 | 0 | 0 |
| 20 | A:A:17 | A:A:18 | 93.61 | 95.31 | 1.78365 | 0 | 0 |
| 21 | A:A:20 | A:A:21 | 100 | 100 | -0 | 0 | 0 |
| 22 | A:A:21 | A:A:22 | 100 | 100 | -0 | 0 | 0 |
| 23 | A:A:22 | A:A:23 | 100 | 100 | -0 | 0 | 0 |
| 24 | A:A:22 | A:A:71 | 56.64 | 41.01 | -27.5953 | 0 | 0 |
| 25 | A:A:23 | A:A:24 | 98.03 | 92.89 | -5.24329 | 0 | 0 |
| 26 | A:A:26 | A:A:27 | 83.22 | 69.48 | -16.5105 | 0 | 0 |
| 27 | A:A:27 | A:A:28 | 100 | 99.93 | -0.0699997 | 0 | 0 |
| 28 | A:A:28 | A:A:29 | 81.15 | 88.1 | 7.88876 | 0 | 0 |
| 29 | A:A:29 | A:A:30 | 100 | 100 | -0 | 0 | 0 |
| 30 | A:A:30 | A:A:31 | 99.97 | 100 | 0.0299988 | 0 | 0 |
| 31 | A:A:31 | A:A:32 | 78.65 | 89.18 | 11.8076 | 0 | 0 |
| 32 | A:A:31 | A:A:36 | 53.53 | 75.75 | 29.3333 | 0 | 0 |
| 33 | A:A:35 | A:A:36 | 100 | 100 | -0 | 0 | 0 |
| 34 | A:A:35 | A:A:58 | 89.13 | 52.03 | -41.6246 | 0 | 0 |
| 35 | A:A:36 | A:A:37 | 100 | 100 | -0 | 0 | 0 |
| 36 | A:A:37 | A:A:38 | 98.26 | 97.49 | -0.78364 | 0 | 0 |
| 37 | A:A:40 | A:A:41 | 100 | 100 | -0 | 0 | 0 |
| 38 | A:A:41 | A:A:42 | 100 | 100 | -0 | 0 | 0 |
| 39 | A:A:42 | A:A:43 | 99.47 | 99.61 | 0.140548 | 0 | 0 |
| 40 | A:A:42 | A:A:47 | 89.55 | 71.76 | -19.866 | 0 | 0 |
| 41 | A:A:47 | A:A:48 | 97.18 | 97.58 | 0.409922 | 0 | 0 |
| 42 | A:A:48 | A:A:49 | 86.98 | 90.94 | 4.35452 | 0 | 0 |
| 43 | A:A:49 | A:A:51 | 100 | 100 | -0 | 0 | 0 |
| 44 | A:A:51 | A:A:52 | 100 | 100 | -0 | 0 | 0 |
| 45 | A:A:52 | A:A:53 | 67.55 | 35.33 | -47.698 | 0 | 0 |
| 46 | A:A:52 | A:A:89 | 57.65 | 58.72 | 1.82221 | 0 | 0 |
| 47 | A:A:53 | A:A:54 | 100 | 100 | -0 | 0 | 0 |
| 48 | A:A:53 | A:A:56 | 74.84 | 62.86 | -16.0075 | 0 | 0 |
| 49 | A:A:56 | A:A:91 | 94.54 | 100 | 5.46 | 0 | 0 |
| 50 | A:A:57 | A:A:58 | 100 | 100 | -0 | 0 | 0 |
| 51 | A:A:57 | A:A:59 | 76.83 | 57.22 | -25.5239 | 0 | 0 |
| 52 | A:A:57 | A:A:90 | 59.56 | 33.11 | -44.409 | 0 | 0 |
| 53 | A:A:58 | A:A:59 | 99.84 | 99.86 | 0.0200323 | 0 | 0 |
| 54 | A:A:61 | A:A:62 | 99.99 | 100 | 0.0100021 | 0 | 0 |
| 55 | A:A:61 | A:A:64 | 99.71 | 99.82 | 0.110199 | 0 | 0 |
| 56 | A:A:64 | A:A:65 | 100 | 100 | -0 | 0 | 0 |
| 57 | A:A:65 | A:A:66 | 91.58 | 85.93 | -6.16947 | 0 | 0 |
| 58 | A:A:66 | A:A:67 | 91.3 | 95.58 | 4.47792 | 0 | 0 |
| 59 | A:A:70 | A:A:71 | 99.99 | 100 | 0.0100021 | 0 | 0 |
| 60 | A:A:71 | A:A:72 | 99.74 | 100 | 0.260002 | 0 | 0 |
| 61 | A:A:72 | A:A:73 | 99.72 | 100 | 0.279999 | 0 | 0 |
| 62 | A:A:75 | A:A:76 | 100 | 99.99 | -0.0100021 | 0 | 0 |
| 63 | A:A:76 | A:A:77 | 100 | 100 | -0 | 0 | 0 |
| 64 | A:A:76 | A:A:79 | 55.58 | 32.1 | -42.2454 | 0 | 0 |
| 65 | A:A:76 | A:A:80 | 54.99 | 63.78 | 13.7817 | 0 | 0 |
| 66 | A:A:77 | A:A:78 | 99.41 | 99.12 | -0.291722 | 0 | 0 |
| 67 | A:A:78 | A:A:79 | 98.4 | 95.65 | -2.79472 | 0 | 0 |
| 68 | A:A:79 | A:A:80 | 90.1 | 97.12 | 7.22818 | 0 | 0 |
| 69 | A:A:80 | A:A:81 | 100 | 100 | -0 | 0 | 0 |
| 70 | A:A:83 | A:A:84 | 92.1 | 99.49 | 7.42788 | 0 | 0 |
| 71 | A:A:84 | A:A:85 | 100 | 100 | -0 | 0 | 0 |
| 72 | A:A:85 | A:A:86 | 77.98 | 59.62 | -23.5445 | 0 | 0 |
| 73 | A:A:86 | A:A:87 | 99.93 | 99.93 | -0 | 0 | 0 |
| 74 | A:A:88 | A:A:89 | 99.96 | 99.96 | -0 | 0 | 0 |
| 75 | A:A:90 | A:A:91 | 99.99 | 99.99 | -0 | 0 | 0 |
| 76 | A:A:93 | A:A:94 | 83.91 | 64.71 | -22.8817 | 0 | 0 |
| 77 | A:A:10 | A:A:9 | 11.1 | 13.87 | 19.9712 | 0 | 0 |
| 78 | A:A:13 | A:A:83 | 40.01 | 33.73 | -15.6961 | 0 | 0 |
| 79 | A:A:23 | A:A:36 | 27.05 | 25.42 | -6.02588 | 0 | 0 |
| 80 | A:A:30 | A:A:33 | 30.16 | 10.48 | -65.252 | 0 | 0 |
| 81 | A:A:91 | A:A:92 | 20.33 | 0.08 | -99.6065 | 0 | 0 |
| 82 | A:A:49 | A:A:50 | 24.27 | 24.14 | -0.535645 | 0 | 0 |
| 83 | A:A:81 | A:A:82 | 0.77 | 0.6 | -22.0779 | 0 | 0 |
| 84 | A:A:61 | A:A:63 | 0 | 0 | 0 | 0 | 0 |
| 85 | A:A:42 | A:A:44 | 0 | 0 | 0 | 0 | 0 |
| 86 | A:A:54 | A:A:55 | 0.09 | 0.14 | 35.7143 | 0 | 0 |
| 87 | A:A:18 | A:A:19 | 0 | 0 | 0 | 0 | 0 |
| 88 | A:A:74 | A:A:75 | 1.51 | 1.65 | 8.48485 | 0 | 0 |
| 89 | A:A:60 | A:A:61 | 0.09 | 0.17 | 47.0588 | 0 | 0 |
| 90 | A:A:69 | A:A:70 | 0.03 | 0 | -100 | 0 | 0 |
| 91 | A:A:67 | A:A:68 | 0.22 | 3.24 | 93.2099 | 0 | 0 |
| 92 | A:A:21 | A:A:39 | 0 | 0 | 0 | 0 | 0 |
| 93 | A:A:12 | A:A:13 | 0 | 0 | 0 | 0 | 0 |
| 94 | A:A:60 | A:A:88 | 0.04 | 0.56 | 92.8571 | 0 | 0 |
| 95 | A:A:46 | A:A:47 | 0 | 0 | 0 | 0 | 0 |
| 96 | A:A:34 | A:A:36 | 17.83 | 25.14 | 29.0772 | 0 | 0 |
| 97 | A:A:11 | A:A:18 | 9.05 | 3.16 | -65.0829 | 0 | 0 |
| 98 | A:A:25 | A:A:35 | 0 | 0 | 0 | 0 | 0 |
| 99 | A:A:45 | A:A:48 | 0 | 0 | 0 | 0 | 0 |
| 100 | A:A:53 | A:A:94 | 16.4 | 6.68 | -59.2683 | 0 | 0 |
| 101 | A:A:11 | A:A:85 | 45.26 | 64.18 | 29.4796 | 0 | 0 |
| 102 | A:A:13 | A:A:16 | 43.78 | 56.17 | 22.058 | 0 | 0 |
| 103 | A:A:20 | B:B:8 | 0 | 88.22 | 100 | 0 | 0 |
| 104 | A:A:22 | B:B:6 | 0 | 96.5 | 100 | 0 | 0 |
| 105 | A:A:36 | A:A:57 | 38.69 | 60.54 | 36.0918 | 0 | 0 |
| 106 | A:A:51 | A:A:91 | 0.85 | 85.04 | 99.0005 | 0 | 0 |
| 107 | A:A:71 | A:A:75 | 17.44 | 74.5 | 76.5906 | 0 | 0 |
| 108 | A:A:71 | B:B:4 | 0 | 96.81 | 100 | 0 | 0 |
| 109 | A:A:71 | B:B:5 | 0 | 52.14 | 100 | 0 | 0 |
| 110 | B:B:3 | B:B:4 | 0 | 55.13 | 100 | 0 | 0 |
| 111 | B:B:4 | B:B:5 | 0 | 91.48 | 100 | 0 | 0 |
| 112 | B:B:5 | B:B:6 | 0 | 100 | 100 | 0 | 0 |
| 113 | A:A:17 | B:B:8 | 0 | 79.42 | 100 | 0 | 0 |
| 114 | A:A:56 | A:A:57 | 75.69 | 52.95 | -30.0436 | 0 | 0 |
| 115 | A:A:57 | A:A:67 | 5.98 | 38.02 | 84.2714 | 0 | 0 |
| 116 | A:A:34 | A:A:57 | 16.54 | 22.43 | 26.2595 | 0 | 0 |
| 117 | B:B:6 | B:B:7 | 0 | 0 | 0 | 0 | 0 |
| 118 | A:A:24 | A:A:25 | 0 | 0 | 0 | 0 | 0 |
| 119 | A:A:45 | A:A:46 | 0 | 0 | 0 | 0 | 0 |
| 120 | A:A:4 | A:A:93 | 3.78 | 11.01 | 65.6676 | 0 | 0 |
Index: link id, click on each number to highlight the corresponding link in the 3D visualization.
Node1 Node2: the two nodes of the corresponding link.
Net1 & Net2 Freq: the trajectory frequency of this link or 0 if the link is not present.
Freq Pert:trajectory frequency perturbation.
Value1, Value2: these columns report the values present in the user submitted external values file (if any).
| Index | Hub | Owner | 3lnx Freq | 3lny Freq | 3lnx Num Of Links | 3lny Num Of Links | Value |
|---|---|---|---|---|---|---|---|
| 1 | A:A:9 | 3lnx | 30.39 | 40.28 | 3 | 3 | 0 |
| 2 | A:A:13 | 3lnx | 67.61 | 47.73 | 5 | 4 | 0 |
| 3 | A:A:53 | 3lnx | 50 | 5.7 | 4 | 2 | 0 |
| 4 | A:A:61 | 3lnx | 97.36 | 76.82 | 4 | 3 | 0 |
| 5 | A:A:76 | 3lnx | 38.9 | 36.19 | 4 | 3 | 0 |
| 6 | A:A:7 | Shared | 74.55 | 63.45 | 5 | 4 | 0 |
| 7 | A:A:36 | Shared | 83.12 | 86.53 | 5 | 4 | 0 |
| 8 | A:A:42 | Shared | 82.67 | 64.08 | 4 | 4 | 0 |
| 9 | A:A:86 | Shared | 49.8 | 46.1 | 4 | 4 | 0 |
| 10 | A:A:91 | Shared | 96.44 | 99.11 | 5 | 5 | 0 |
| 11 | A:A:57 | 3lny | 84 | 76.95 | 4 | 5 | 0 |
| 12 | A:A:71 | 3lny | 21.46 | 91.54 | 3 | 5 | 0 |
Index: hub id, click on each number to highlight the corresponding hub in the 3D visualization.
Hub: the hub being considered.
Owner: the name of the network the hub belong to or "Shared" if the corresponding hub is present in both networks.
Freq: the trajectory frequency of the corresponding hub in the corresponding network.
Num Of Links: the number of links of the corresponding hub in the corresponding network.
Value: this column reports the value present in the user submitted external values file (if any).
| Index | Hub | 3lnx Freq | 3lny Freq | Freq Pert | Value |
|---|---|---|---|---|---|
| 1 | A:A:7 | 74.55 | 63.45 | 14.8893 | 0 |
| 2 | A:A:9 | 30.39 | 40.28 | -24.5531 | 0 |
| 3 | A:A:13 | 67.61 | 47.73 | 29.4039 | 0 |
| 4 | A:A:36 | 83.12 | 86.53 | -3.94083 | 0 |
| 5 | A:A:42 | 82.67 | 64.08 | 22.487 | 0 |
| 6 | A:A:53 | 50 | 5.7 | 88.6 | 0 |
| 7 | A:A:61 | 97.36 | 76.82 | 21.097 | 0 |
| 8 | A:A:76 | 38.9 | 36.19 | 6.96659 | 0 |
| 9 | A:A:86 | 49.8 | 46.1 | 7.42972 | 0 |
| 10 | A:A:91 | 96.44 | 99.11 | -2.69397 | 0 |
| 11 | A:A:57 | 84 | 76.95 | 8.39285 | 0 |
| 12 | A:A:71 | 21.46 | 91.54 | -76.5567 | 0 |
Index: hub id, click on each number to highlight the corresponding hub in the 3D visualization.
Hub: the hub being considered.
Net1 & Net2 Freq: the trajectory frequency of this link or 0 if the link is not present.
Freq Pert:trajectory frequency perturbation.
Value: this column reports the value present in the user submitted external values file (if any).
| Index | Node1 | Node2 | Owner | 3lnx Recurrency | 3lny Recurrency | 3lnx Hub1? | 3lny Hub1? | 3lnx Hub2? | 3lny Hub2? | Value1 | Value2 |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | A:A:8 | A:A:9 | 3lnx | 22.736 | 6.82426 | No | No | Yes | No | 0 | 0 |
| 2 | A:A:10 | A:A:9 | 3lnx | 21.3391 | 0 | No | No | Yes | No | 0 | 0 |
| 3 | A:A:85 | A:A:86 | 3lnx | 63.8247 | 19.8037 | No | No | Yes | Yes | 0 | 0 |
| 4 | A:A:84 | A:A:85 | 3lnx | 60.5491 | 8.7868 | No | No | No | No | 0 | 0 |
| 5 | A:A:83 | A:A:84 | 3lnx | 57.1773 | 4.4157 | No | No | No | No | 0 | 0 |
| 6 | A:A:13 | A:A:83 | 3lnx | 53.6127 | 0 | Yes | No | No | No | 0 | 0 |
| 7 | A:A:90 | A:A:91 | 3lnx | 100 | 4.4603 | No | No | Yes | Yes | 0 | 0 |
| 8 | A:A:57 | A:A:90 | 3lnx | 97.1098 | 0 | No | Yes | No | No | 0 | 0 |
| 9 | A:A:57 | A:A:58 | 3lnx | 92.8709 | 10.1249 | No | Yes | No | No | 0 | 0 |
| 10 | A:A:35 | A:A:58 | 3lnx | 91.0405 | 4.99554 | No | No | No | No | 0 | 0 |
| 11 | A:A:35 | A:A:36 | 3lnx | 87.7649 | 7.85013 | No | No | Yes | Yes | 0 | 0 |
| 12 | A:A:29 | A:A:30 | 3lnx | 20.183 | 17.6182 | No | No | No | No | 0 | 0 |
| 13 | A:A:7 | A:A:89 | 3lnx | 74.422 | 9.45584 | Yes | Yes | No | No | 0 | 0 |
| 14 | A:A:52 | A:A:89 | 3lnx | 50.4817 | 1.78412 | No | No | No | No | 0 | 0 |
| 15 | A:A:51 | A:A:52 | 3lnx | 59.4412 | 4.72792 | No | No | No | No | 0 | 0 |
| 16 | A:A:42 | A:A:47 | 3lnx | 24.2293 | 17.2168 | Yes | Yes | No | No | 0 | 0 |
| 17 | A:A:53 | A:A:56 | 3lnx | 32.8516 | 11.686 | Yes | No | No | No | 0 | 0 |
| 18 | A:A:52 | A:A:53 | 3lnx | 31.1657 | 0 | No | No | Yes | No | 0 | 0 |
| 19 | A:A:88 | A:A:89 | 3lnx | 37.9576 | 7.4041 | No | No | No | No | 0 | 0 |
| 20 | A:A:60 | A:A:88 | 3lnx | 34.0559 | 2.9884 | No | No | No | No | 0 | 0 |
| 21 | A:A:60 | A:A:61 | 3lnx | 31.6474 | 0 | No | No | Yes | No | 0 | 0 |
| 22 | A:A:61 | A:A:64 | 3lnx | 20.8092 | 11.6414 | Yes | No | No | No | 0 | 0 |
| 23 | A:A:22 | A:A:71 | 3lnx | 23.2659 | 0 | No | No | No | Yes | 0 | 0 |
| 24 | A:A:13 | A:A:81 | 3lnx | 36.4162 | 18.7333 | Yes | No | No | No | 0 | 0 |
| 25 | A:A:7 | A:A:91 | Shared | 88.2948 | 64.7636 | Yes | Yes | Yes | Yes | 0 | 0 |
| 26 | A:A:7 | A:A:8 | Shared | 88.6802 | 36.6191 | Yes | Yes | No | No | 0 | 0 |
| 27 | A:A:8 | A:A:86 | Shared | 63.3911 | 26.0036 | No | No | Yes | Yes | 0 | 0 |
| 28 | A:A:23 | A:A:36 | Shared | 46.869 | 93.8894 | No | No | Yes | Yes | 0 | 0 |
| 29 | A:A:22 | A:A:23 | Shared | 39.9326 | 90.1427 | No | No | No | No | 0 | 0 |
| 30 | A:A:31 | A:A:36 | Shared | 35.4528 | 30.8653 | No | No | Yes | Yes | 0 | 0 |
| 31 | A:A:30 | A:A:31 | Shared | 27.9865 | 24.3979 | No | No | No | No | 0 | 0 |
| 32 | A:A:49 | A:A:51 | Shared | 53.9981 | 38.9384 | No | No | No | No | 0 | 0 |
| 33 | A:A:48 | A:A:49 | Shared | 45.183 | 33.0062 | No | No | No | No | 0 | 0 |
| 34 | A:A:47 | A:A:48 | Shared | 35.8863 | 29.215 | No | No | No | No | 0 | 0 |
| 35 | A:A:56 | A:A:91 | Shared | 34.9229 | 94.2016 | No | No | Yes | Yes | 0 | 0 |
| 36 | A:A:80 | A:A:81 | Shared | 29.9133 | 26.8956 | No | No | No | No | 0 | 0 |
| 37 | A:A:5 | A:A:6 | 3lny | 8.18882 | 23.9072 | No | No | No | No | 0 | 0 |
| 38 | A:A:6 | A:A:7 | 3lny | 12.5723 | 27.7877 | No | No | Yes | Yes | 0 | 0 |
| 39 | A:A:56 | A:A:57 | 3lny | 0 | 98.7065 | No | No | No | Yes | 0 | 0 |
| 40 | A:A:36 | A:A:57 | 3lny | 0 | 100 | Yes | Yes | No | Yes | 0 | 0 |
| 41 | A:A:22 | B:B:6 | 3lny | 0 | 75.5129 | No | No | No | No | 0 | 0 |
| 42 | B:B:5 | B:B:6 | 3lny | 0 | 71.6771 | No | No | No | No | 0 | 0 |
| 43 | A:A:71 | B:B:5 | 3lny | 0 | 62.8011 | No | Yes | No | No | 0 | 0 |
| 44 | A:A:71 | A:A:75 | 3lny | 0 | 49.6878 | No | Yes | No | No | 0 | 0 |
| 45 | A:A:75 | A:A:76 | 3lny | 9.58574 | 44.2462 | No | No | Yes | No | 0 | 0 |
| 46 | A:A:76 | A:A:80 | 3lny | 16.5222 | 32.7386 | Yes | No | No | No | 0 | 0 |
| 47 | A:A:21 | A:A:22 | 3lny | 13.5356 | 25.6467 | No | No | No | No | 0 | 0 |
| 48 | A:A:51 | A:A:91 | 3lny | 0 | 42.5513 | No | No | Yes | Yes | 0 | 0 |
| 49 | A:A:57 | A:A:67 | 3lny | 0 | 30.5085 | No | Yes | No | No | 0 | 0 |
| 50 | A:A:66 | A:A:67 | 3lny | 9.20039 | 24.1302 | No | No | No | No | 0 | 0 |
| 51 | A:A:65 | A:A:66 | 3lny | 13.6802 | 20.0714 | No | No | No | No | 0 | 0 |
Index: link id, click on each number to highlight the corresponding link in the 3D visualization.
Node1 Node2: the two nodes of the corresponding link.
Owner: the name of the network the link belong to or "Shared" if the corresponding link is present in both networks.
Recurrency: the relative recurrency in the filtered pool of shortest paths.
Hub1?, Hub2?: "Yes" if the corresponding node has more than 3 links, otherwise "No".
Value1, Value2: these columns report the values present in the user submitted external values file (if any).
Here, those paths that begin and end at given residue pair(s) or that pass through a residue are used to generate a novel metapath. Such a path filtering is particularly recommended if some information on allosteric residues is available. The selection syntax for a residue pair ("Pairs") is C:C:c534,A:A:?600 (the lower-case "c" stands for cytosine and "?" indicates any non standard amino acid or nucleotide residue). Selection of more than one apical residue pair has the following syntax: C:C:c534,A:A:?600,C:C:c534,A:A:E41. Only one residue can be selected as a tail ("Tail"). For midway residue(s), each residue must be separated by a comma as well e.g. A:A:?600,A:A:E41.
| Index | Node1 | Node2 | Owner | 3lnx Recurrency | 3lny Recurrency | 3lnx Hub1? | 3lny Hub1? | 3lnx Hub2? | 3lny Hub2? | Value1 | Value2 |
|---|
Index: link id, click on each number to highlight the corresponding link in the 3D visualization.
Node1 Node2: the two nodes of the corresponding link.
Owner: the name of the network the link belong to or "Shared" if the corresponding link is present in both networks.
Recurrency: the relative recurrency in the filtered pool of shortest paths.
Hub1?, Hub2?: "Yes" if the corresponding node has more than 3 links, otherwise "No".
Value1, Value2: these columns report the values present in the user submitted external values file (if any).
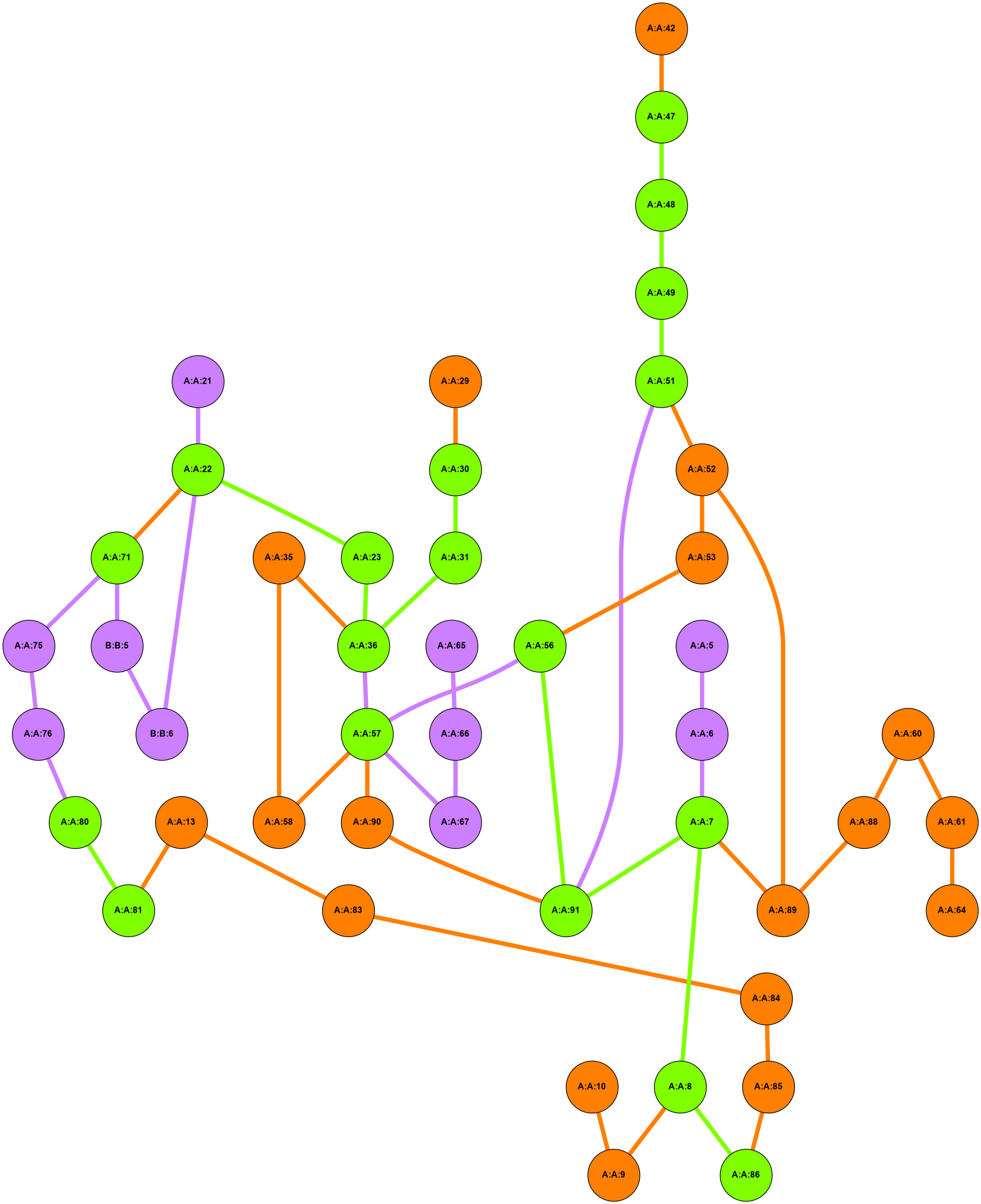
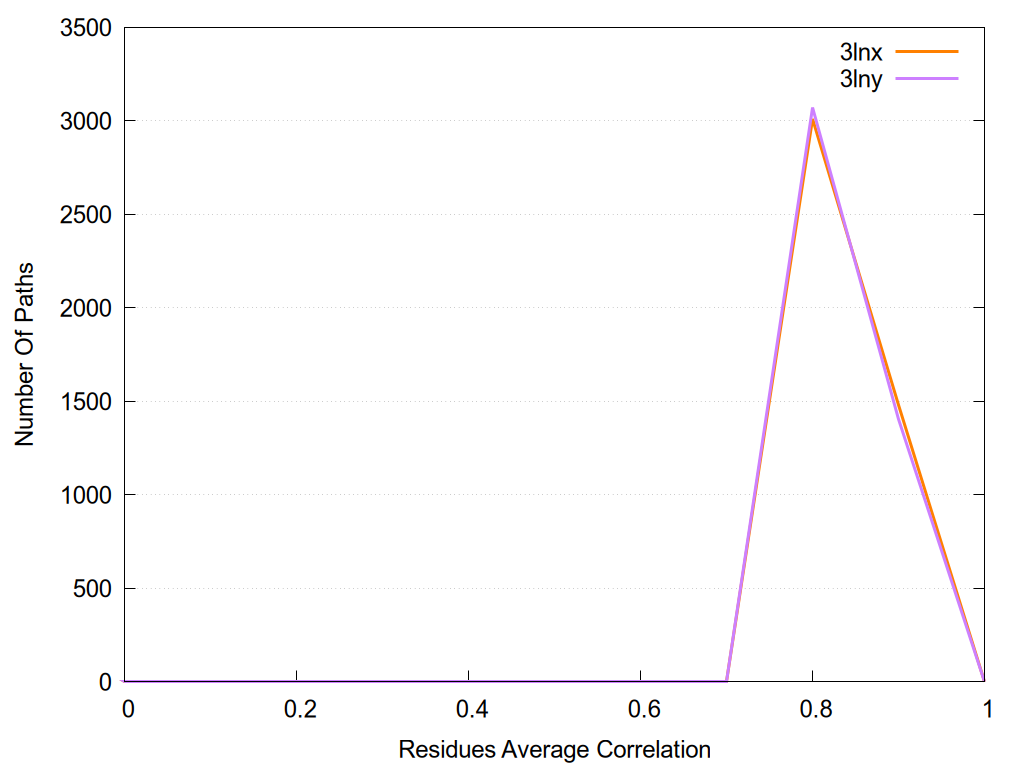
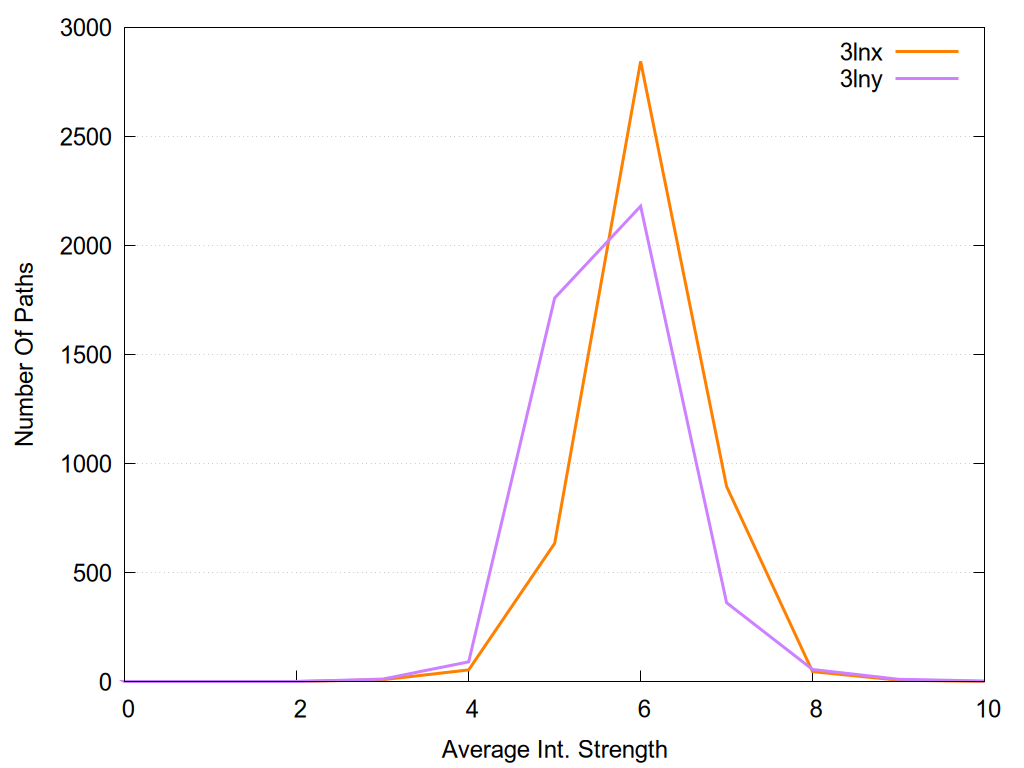
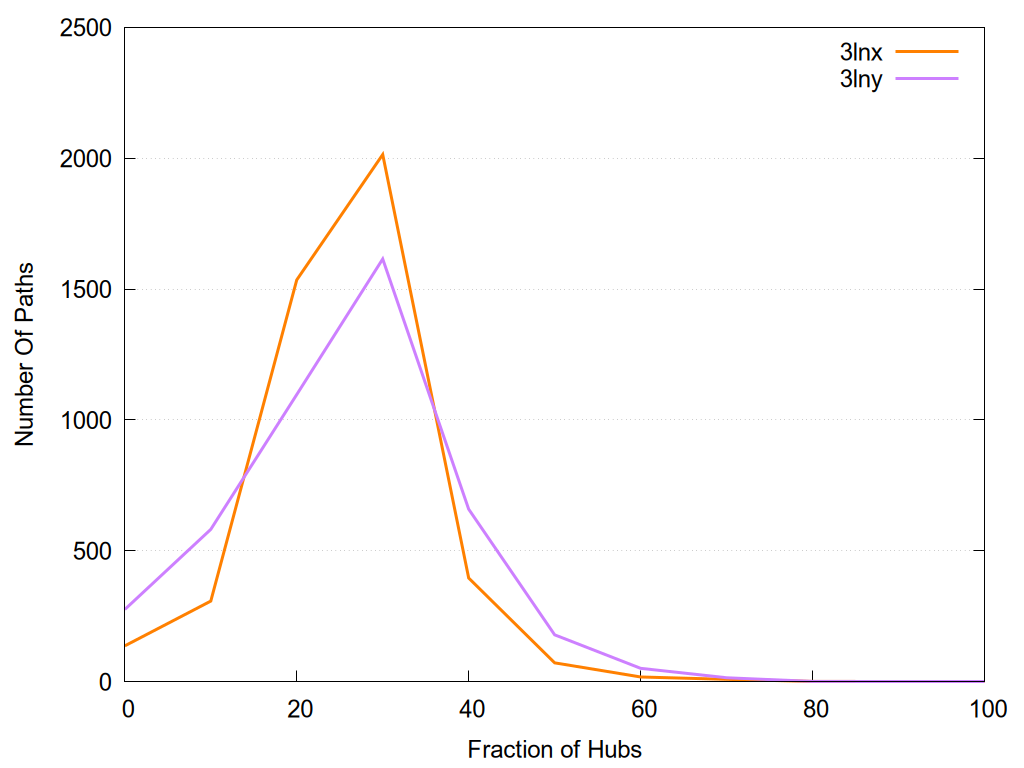
2D representation of the global metapath and histograms of path distribution according to several parameters:
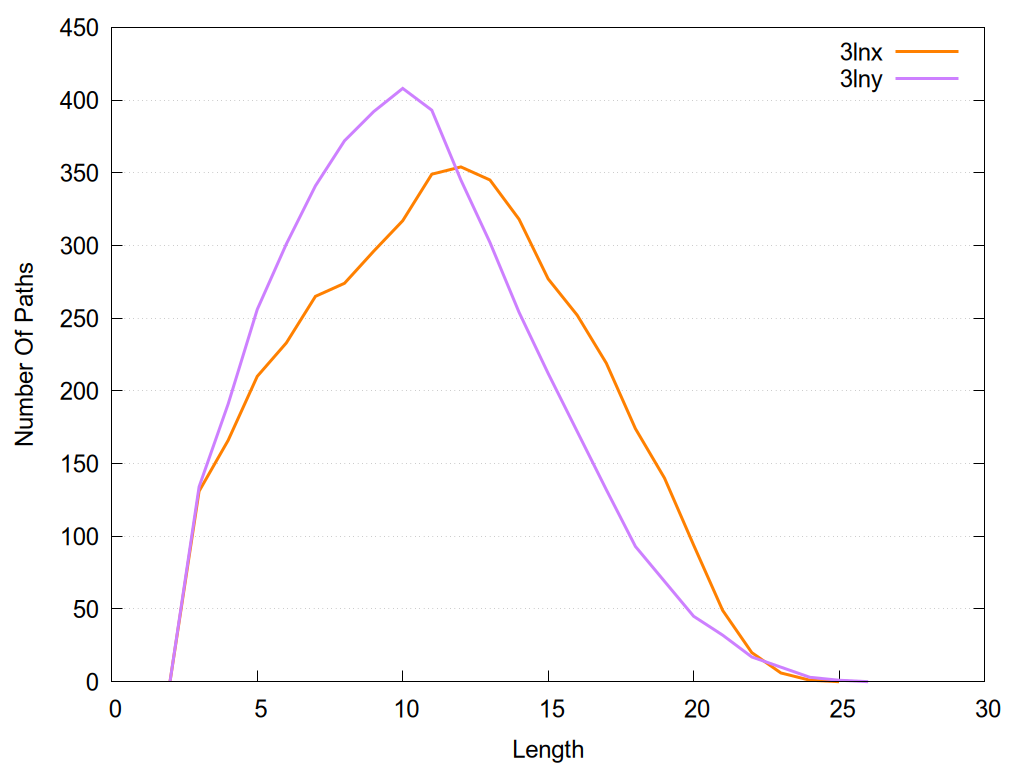
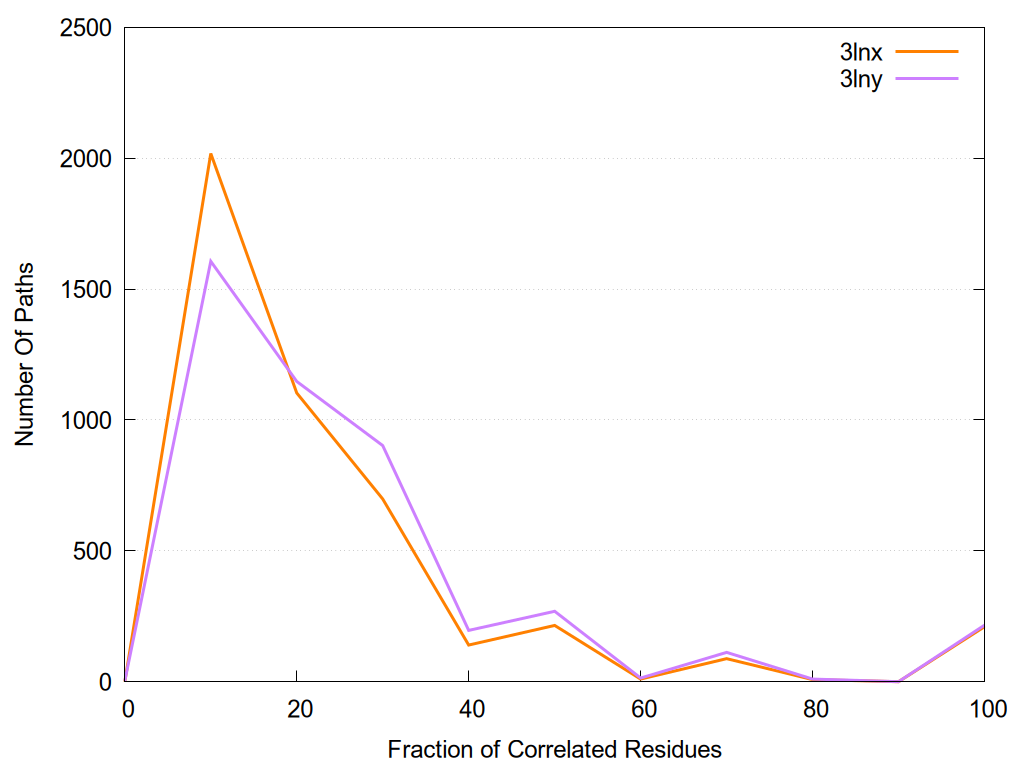
2D representation of the user filetered metapath and histograms of path distribution according to several parameters:






|
|
|
Details about the values in these tables can be found in the corresponding documentation page .
; ; msa not aveilable if you choose not to apply labels ;
| N | Lab | PresentIn | 3lnx | 3lny |
|---|---|---|---|---|
| 1 | A:A:1 | 100.00 | A:A:P1 | A:A:P1 |
| 2 | A:A:2 | 100.00 | A:A:K2 | A:A:K2 |
| 3 | A:A:3 | 100.00 | A:A:P3 | A:A:P3 |
| 4 | A:A:4 | 100.00 | A:A:G4 | A:A:G4 |
| 5 | A:A:5 | 100.00 | A:A:D5 | A:A:D5 |
| 6 | A:A:6 | 100.00 | A:A:I6 | A:A:I6 |
| 7 | A:A:7 | 100.00 | A:A:F7 | A:A:F7 |
| 8 | A:A:8 | 100.00 | A:A:E8 | A:A:E8 |
| 9 | A:A:9 | 100.00 | A:A:V9 | A:A:V9 |
| 10 | A:A:10 | 100.00 | A:A:E10 | A:A:E10 |
| 11 | A:A:11 | 100.00 | A:A:L11 | A:A:L11 |
| 12 | A:A:12 | 100.00 | A:A:A12 | A:A:A12 |
| 13 | A:A:13 | 100.00 | A:A:K13 | A:A:K13 |
| 14 | A:A:14 | 100.00 | A:A:N14 | A:A:N14 |
| 15 | A:A:15 | 100.00 | A:A:D15 | A:A:D15 |
| 16 | A:A:16 | 100.00 | A:A:N16 | A:A:N16 |
| 17 | A:A:17 | 100.00 | A:A:S17 | A:A:S17 |
| 18 | A:A:18 | 100.00 | A:A:L18 | A:A:L18 |
| 19 | A:A:19 | 100.00 | A:A:G19 | A:A:G19 |
| 20 | A:A:20 | 100.00 | A:A:I20 | A:A:I20 |
| 21 | A:A:21 | 100.00 | A:A:S21 | A:A:S21 |
| 22 | A:A:22 | 100.00 | A:A:V22 | A:A:V22 |
| 23 | A:A:23 | 100.00 | A:A:T23 | A:A:T23 |
| 24 | A:A:24 | 100.00 | A:A:G24 | A:A:G24 |
| 25 | A:A:25 | 100.00 | A:A:G25 | A:A:G25 |
| 26 | A:A:26 | 100.00 | A:A:V26 | A:A:V26 |
| 27 | A:A:27 | 100.00 | A:A:N27 | A:A:N27 |
| 28 | A:A:28 | 100.00 | A:A:T28 | A:A:T28 |
| 29 | A:A:29 | 100.00 | A:A:S29 | A:A:S29 |
| 30 | A:A:30 | 100.00 | A:A:V30 | A:A:V30 |
| 31 | A:A:31 | 100.00 | A:A:R31 | A:A:R31 |
| 32 | A:A:32 | 100.00 | A:A:H32 | A:A:H32 |
| 33 | A:A:33 | 100.00 | A:A:G33 | A:A:G33 |
| 34 | A:A:34 | 100.00 | A:A:G34 | A:A:G34 |
| 35 | A:A:35 | 100.00 | A:A:I35 | A:A:I35 |
| 36 | A:A:36 | 100.00 | A:A:Y36 | A:A:Y36 |
| 37 | A:A:37 | 100.00 | A:A:V37 | A:A:V37 |
| 38 | A:A:38 | 100.00 | A:A:K38 | A:A:K38 |
| 39 | A:A:39 | 100.00 | A:A:A39 | A:A:A39 |
| 40 | A:A:40 | 100.00 | A:A:V40 | A:A:V40 |
| 41 | A:A:41 | 100.00 | A:A:I41 | A:A:I41 |
| 42 | A:A:42 | 100.00 | A:A:P42 | A:A:P42 |
| 43 | A:A:43 | 100.00 | A:A:Q43 | A:A:Q43 |
| 44 | A:A:44 | 100.00 | A:A:G44 | A:A:G44 |
| 45 | A:A:45 | 100.00 | A:A:A45 | A:A:A45 |
| 46 | A:A:46 | 100.00 | A:A:A46 | A:A:A46 |
| 47 | A:A:47 | 100.00 | A:A:E47 | A:A:E47 |
| 48 | A:A:48 | 100.00 | A:A:S48 | A:A:S48 |
| 49 | A:A:49 | 100.00 | A:A:D49 | A:A:D49 |
| 50 | A:A:50 | 100.00 | A:A:G50 | A:A:G50 |
| 51 | A:A:51 | 100.00 | A:A:R51 | A:A:R51 |
| 52 | A:A:52 | 100.00 | A:A:I52 | A:A:I52 |
| 53 | A:A:53 | 100.00 | A:A:H53 | A:A:H53 |
| 54 | A:A:54 | 100.00 | A:A:K54 | A:A:K54 |
| 55 | A:A:55 | 100.00 | A:A:G55 | A:A:G55 |
| 56 | A:A:56 | 100.00 | A:A:D56 | A:A:D56 |
| 57 | A:A:57 | 100.00 | A:A:R57 | A:A:R57 |
| 58 | A:A:58 | 100.00 | A:A:V58 | A:A:V58 |
| 59 | A:A:59 | 100.00 | A:A:L59 | A:A:L59 |
| 60 | A:A:60 | 100.00 | A:A:A60 | A:A:A60 |
| 61 | A:A:61 | 100.00 | A:A:V61 | A:A:V61 |
| 62 | A:A:62 | 100.00 | A:A:N62 | A:A:N62 |
| 63 | A:A:63 | 100.00 | A:A:G63 | A:A:G63 |
| 64 | A:A:64 | 100.00 | A:A:V64 | A:A:V64 |
| 65 | A:A:65 | 100.00 | A:A:S65 | A:A:S65 |
| 66 | A:A:66 | 100.00 | A:A:L66 | A:A:L66 |
| 67 | A:A:67 | 100.00 | A:A:E67 | A:A:E67 |
| 68 | A:A:68 | 100.00 | A:A:G68 | A:A:G68 |
| 69 | A:A:69 | 100.00 | A:A:A69 | A:A:A69 |
| 70 | A:A:70 | 100.00 | A:A:T70 | A:A:T70 |
| 71 | A:A:71 | 100.00 | A:A:H71 | A:A:H71 |
| 72 | A:A:72 | 100.00 | A:A:K72 | A:A:K72 |
| 73 | A:A:73 | 100.00 | A:A:Q73 | A:A:Q73 |
| 74 | A:A:74 | 100.00 | A:A:A74 | A:A:A74 |
| 75 | A:A:75 | 100.00 | A:A:V75 | A:A:V75 |
| 76 | A:A:76 | 100.00 | A:A:E76 | A:A:E76 |
| 77 | A:A:77 | 100.00 | A:A:T77 | A:A:T77 |
| 78 | A:A:78 | 100.00 | A:A:L78 | A:A:L78 |
| 79 | A:A:79 | 100.00 | A:A:R79 | A:A:R79 |
| 80 | A:A:80 | 100.00 | A:A:N80 | A:A:N80 |
| 81 | A:A:81 | 100.00 | A:A:T81 | A:A:T81 |
| 82 | A:A:82 | 100.00 | A:A:G82 | A:A:G82 |
| 83 | A:A:83 | 100.00 | A:A:Q83 | A:A:Q83 |
| 84 | A:A:84 | 100.00 | A:A:V84 | A:A:V84 |
| 85 | A:A:85 | 100.00 | A:A:V85 | A:A:V85 |
| 86 | A:A:86 | 100.00 | A:A:H86 | A:A:H86 |
| 87 | A:A:87 | 100.00 | A:A:L87 | A:A:L87 |
| 88 | A:A:88 | 100.00 | A:A:L88 | A:A:L88 |
| 89 | A:A:89 | 100.00 | A:A:L89 | A:A:L89 |
| 90 | A:A:90 | 100.00 | A:A:E90 | A:A:E90 |
| 91 | A:A:91 | 100.00 | A:A:K91 | A:A:K91 |
| 92 | A:A:92 | 100.00 | A:A:G92 | A:A:G92 |
| 93 | A:A:93 | 100.00 | A:A:Q93 | A:A:Q93 |
| 94 | A:A:94 | 100.00 | A:A:S94 | A:A:S94 |
| 95 | B:B:3 | 50.00 | - | B:B:E3 |
| 96 | B:B:4 | 50.00 | - | B:B:Q4 |
| 97 | B:B:5 | 50.00 | - | B:B:V5 |
| 98 | B:B:6 | 50.00 | - | B:B:S6 |
| 99 | B:B:7 | 50.00 | - | B:B:A7 |
| 100 | B:B:8 | 50.00 | - | B:B:V8 |
You can download a compressed (zip) file with all submitted structures, 3D outputs (as PyMol and VMD scripts) and numerical data files (as csv and plain text files). The zip bundle also contains an output manual explaining the meaning of all files and numerical data.
